在临床医学实践中,生存分析是一种重要的研究方法。生存分析是研究生存时间的分布规律,以及生存时间和相关因素之间关系的一种统计分析方法。它主要的目的是对生存率和时间进行建模,计算患者在特定时间段内生存的概率,主要用于评估治疗的效果和疾病的危险程度。
生存分析的方法一般可以分为三类:
参数法:知道生存时间的分布模型,然后根据数据来估计模型参数,最后以分布模型来计算生存率 。
非参数法:不需要生存时间分布,根据样本统计量来估计生存率,常见方法Kaplan-Meier法(乘积极限法)、Log-rank检验等
半参数法:也不需要生存时间的分布,但最终是通过模型来评估影响生存率的因素,最为常见的是Cox回归模型 。
生存曲线(survival curve)则是将每个时间点的生存率连接在一起的曲线,一般随访时间为X轴,生存率为Y轴;曲线平滑则说明高生存率,反之则低生存率;中位生存率(median survival time)越长,则说明预后较好。本文主要介绍生存曲线的绘制。
安装与加载
install.packages("survminer")
library("survminer")ggsurvplot绘制生存曲线
#调用survival包
require("survival")
#survival包自带肺癌数据集:lung,查看数据样式
head(lung)
> head(lung)
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0
6 12 1022 1 74 1 1 50 80 513 0绘制基础版单一分组的生存曲线
#通过survival包的Surv函数对时间及生存状态进行拟合(检验: Kaplan-Meier法) fit1 <- survfit(Surv(time, status) ~ 1, data = lung) #绘制基础版生存曲线 ggsurvplot(fit, palette = "#2E9FDF") #自定义修改颜色
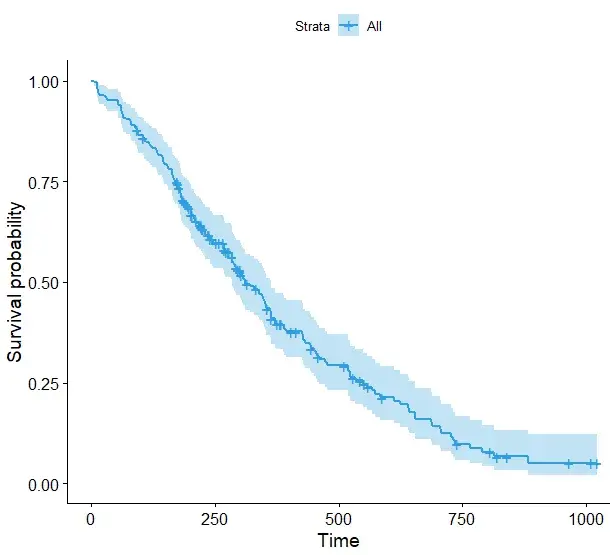
绘制两组的生存曲线
#以性别特征进行分组为例 #绘制基础图形 fit <- survfit(Surv(time, status) ~ sex, data = lung) ggsurvplot(fit, data = lung)
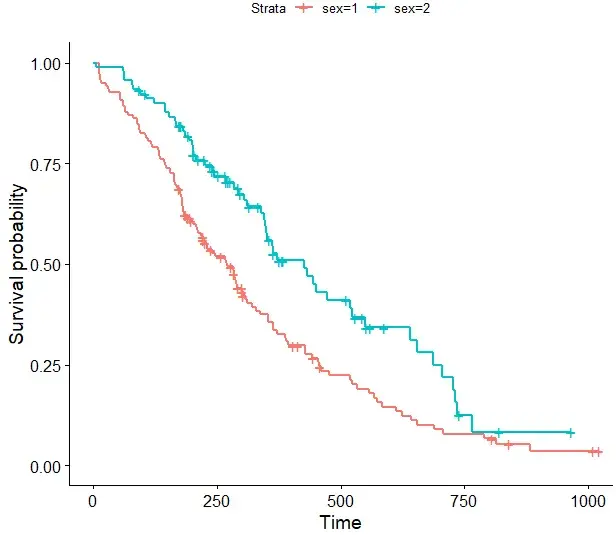
#更改删失点形状及大小,默认为"+", 可选"|" ggsurvplot(fit, data = lung, censor.shape="|", censor.size = 4)
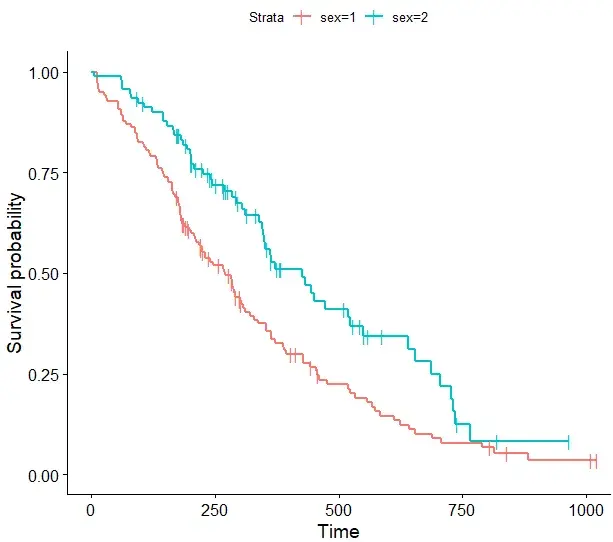
# 使用啤酒调色板“Dark2” ggsurvplot(fit, linetype = "strata", conf.int = TRUE, pval = TRUE, palette = "Dark2")
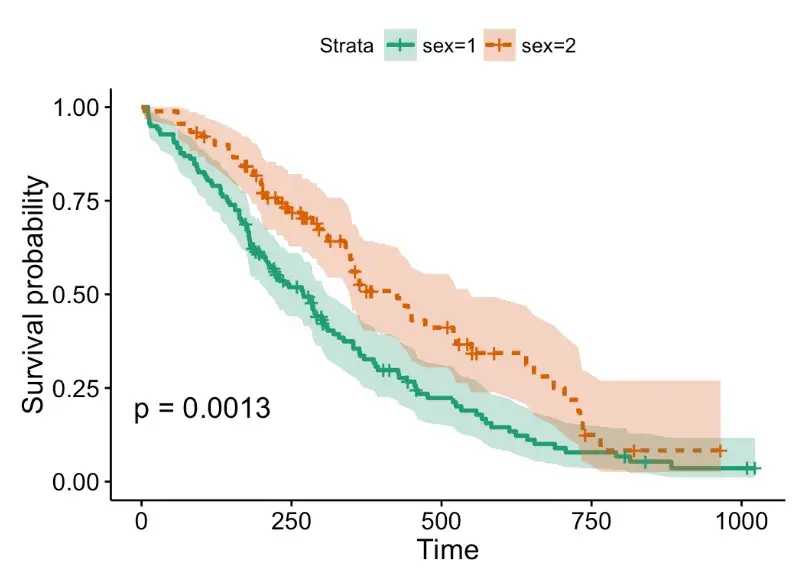
# 使用灰色调色板 ggsurvplot(fit, linetype = "strata", conf.int = TRUE, pval = TRUE, palette = "grey")
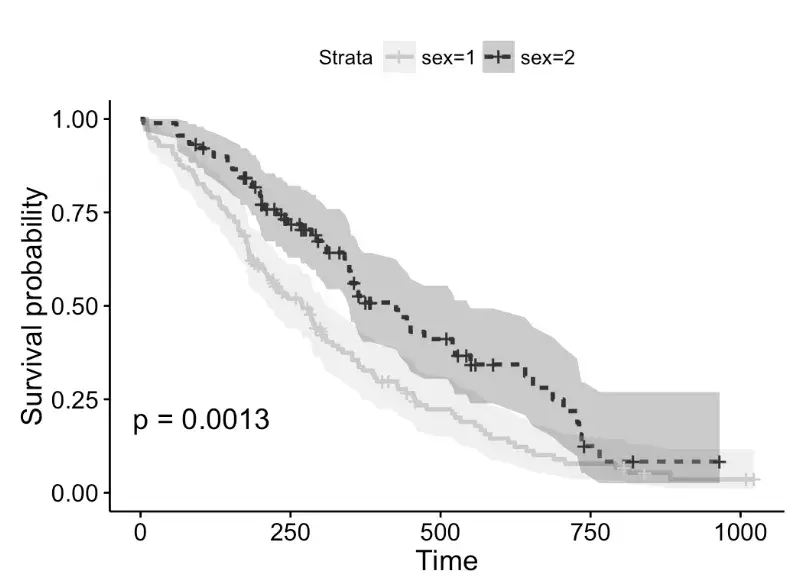
#还支持累计风险曲线绘制 ggsurvplot(fit, data = lung, conf.int = TRUE, # 是否需要增加置信区间 fun = "cumhaz") # cumhaz函数绘制累计风险曲线
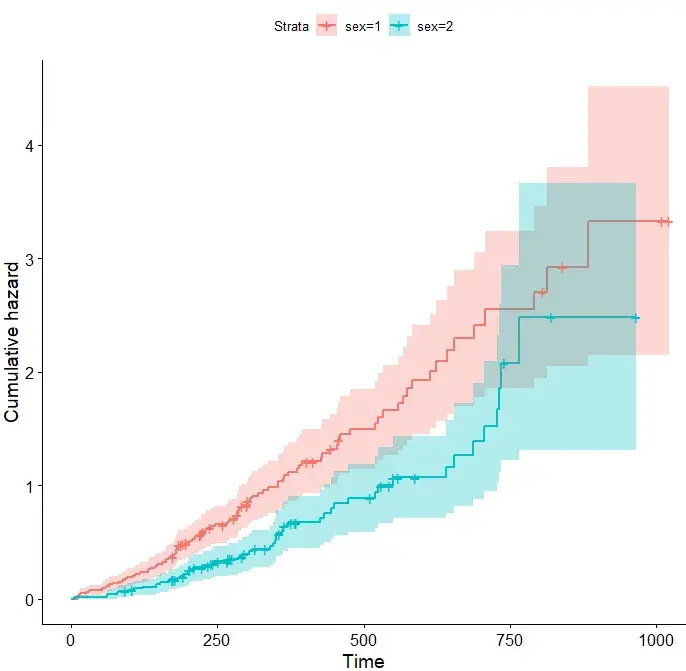
# 指定数据,添加总患者生存曲线 ggsurvplot(fit, # 分组拟合对象 data = lung, # 使用的变量数据集来源 conf.int = TRUE, # 是否显示显示置信区间 pval = TRUE, # 通过 pval 添加P值 surv.median.line = "hv", # surv.median.line添加中位生存时间线 add.all = TRUE) # add.all 添加总患者生存曲线
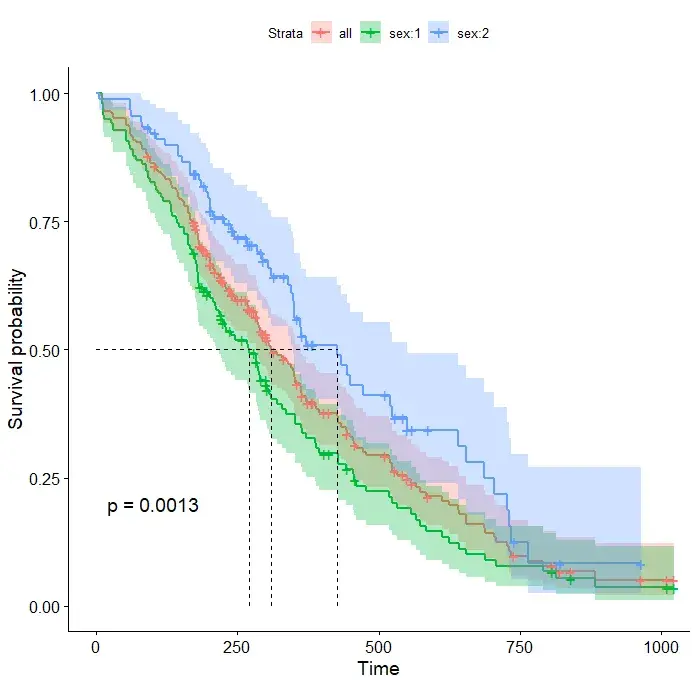
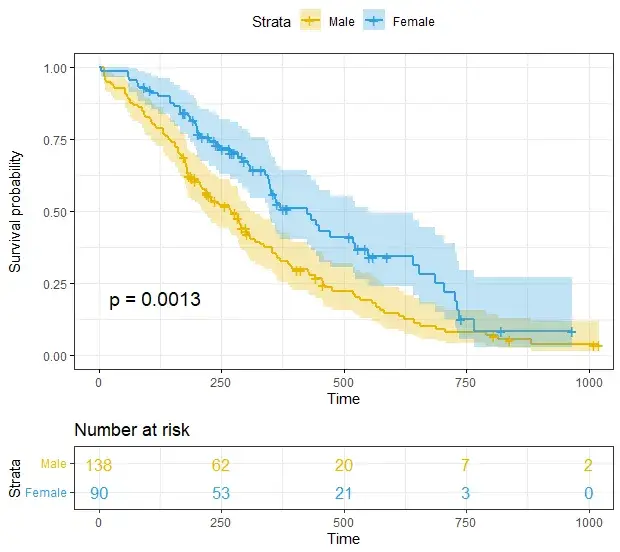
# 通过修改函数接口,制定个性化的生存曲线
ggsurvplot(
fit,
data = lung,
size = 1, # 线条粗细
palette =
c("#E7B800", "#2E9FDF"),# 设置分组颜色
conf.int = TRUE, # conf.int 函数添加置信区间
pval = TRUE, # p值函数添加显著性
risk.table = TRUE, # 添加风险表个绘制
risk.table.col = "strata",# 分线表颜色
legend.labs =
c("Male", "Female"), # 添加对应图例标签
risk.table.height = 0.25, # 生存曲线图下所有生存表的高度,数值0-1之间
ggtheme = theme_bw() # 是否添加图主题
)
# 修改参数,更加个性化的生存曲线图绘制 ggsurvplot( fit, data = lung, risk.table = TRUE, pval = TRUE, conf.int = TRUE, xlim = c(0,500), # xlim, ylim 函数指定x轴和y轴的范围 xlab = "Time in days", # xlab, ylab 这两个函数分别指x轴和y轴标签 break.time.by = 100, # 设定坐标轴刻度间距大小,按需设置 ggtheme = theme_light(), risk.table.y.text.col = T, # 分线表y轴文字字体颜色,可修改 risk.table.y.text = FALSE # 风险表y轴展示条形图例 )
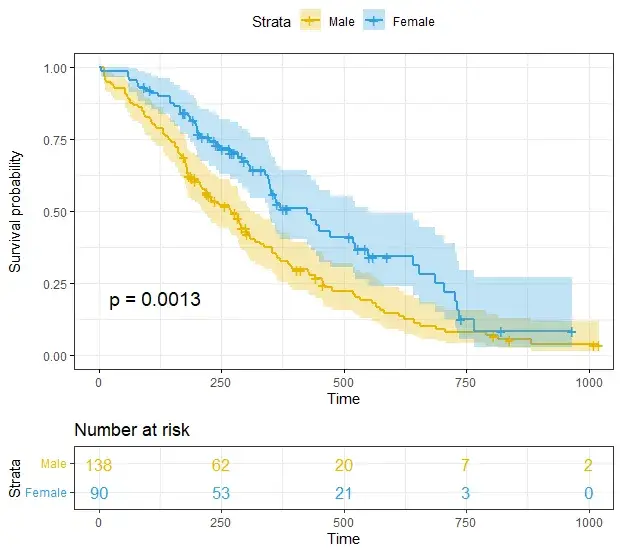
# Uber定制生存曲线
ggsurv <- ggsurvplot(
fit,
data = lung,
risk.table = TRUE,
pval = TRUE,
conf.int = TRUE,
palette = c("#E7B800", "#2E9FDF"),
xlim = c(0,500),
xlab = "Time in days",
break.time.by = 100,
ggtheme = theme_light(),
risk.table.y.text.col = T,
risk.table.height = 0.25,
risk.table.y.text = FALSE,
ncensor.plot = TRUE,
ncensor.plot.height = 0.25,
conf.int.style = "step",
surv.median.line = "hv",
legend.labs =
c("Male", "Female")
)
ggsurv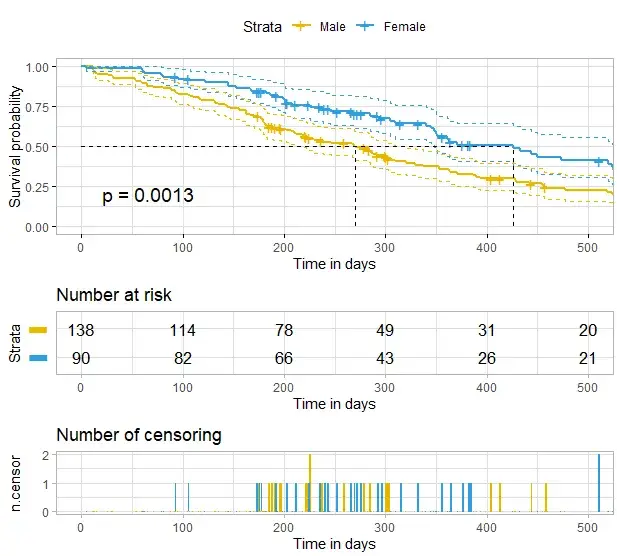
# 引入customize_labels 函数进行更高级的定制,这种函数可作为source对象,加在之后可以直接绘制相应的生存曲线
customize_labels <- function (p, font.title = NULL,
font.subtitle = NULL, font.caption = NULL,
font.x = NULL, font.y = NULL, font.xtickslab = NULL, font.ytickslab = NULL)
{
original.p <- p
if(is.ggplot(original.p)) list.plots <- list(original.p)
else if(is.list(original.p)) list.plots <- original.p
else stop("Can't handle an object of class ", class (original.p))
.set_font <- function(font){
font <- ggpubr:::.parse_font(font)
ggtext::element_markdown (size = font$size, face = font$face, colour = font$color)
}
for(i in 1:length(list.plots)){
p <- list.plots[[i]]
if(is.ggplot(p)){
if (!is.null(font.title)) p <- p + theme(plot.title = .set_font(font.title))
if (!is.null(font.subtitle)) p <- p + theme(plot.subtitle = .set_font(font.subtitle))
if (!is.null(font.caption)) p <- p + theme(plot.caption = .set_font(font.caption))
if (!is.null(font.x)) p <- p + theme(axis.title.x = .set_font(font.x))
if (!is.null(font.y)) p <- p + theme(axis.title.y = .set_font(font.y))
if (!is.null(font.xtickslab)) p <- p + theme(axis.text.x = .set_font(font.xtickslab))
if (!is.null(font.ytickslab)) p <- p + theme(axis.text.y = .set_font(font.ytickslab))
list.plots[[i]] <- p
}
}
if(is.ggplot(original.p)) list.plots[[1]]
else list.plots
}
#用户自定义生存曲线
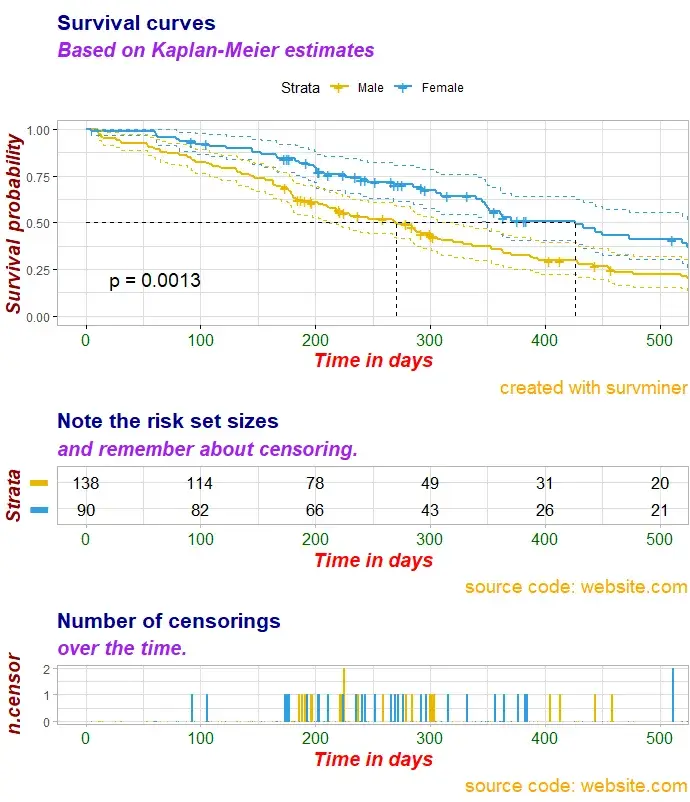
ggsurv$plot <- ggsurv$plot + labs(
title = "Survival curves", #主标题,可改写
subtitle = "Based on Kaplan-Meier estimates", #添加副标题
caption = "created with survminer" # 是否需要说明
)
ggsurv$plot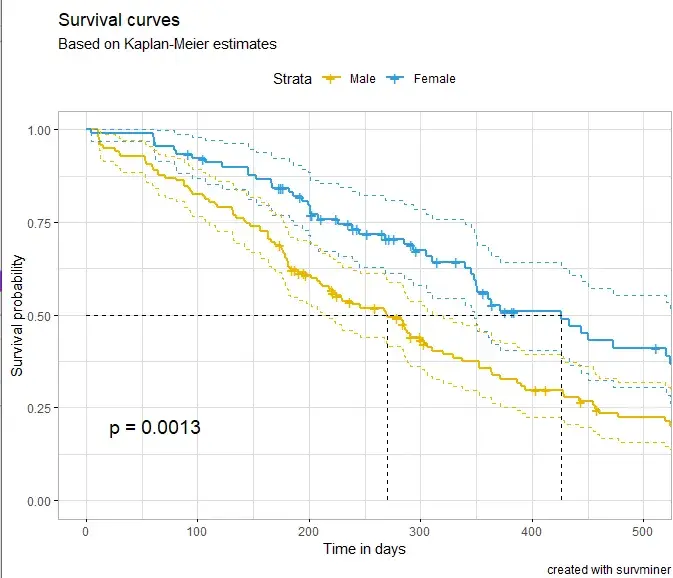
# 单独绘制风险表格 ggsurv$table <- ggsurv$table + labs( title = "Note the risk set sizes", subtitle = "and remember about censoring.", caption = "source code: website.com" ) ggsurv$table
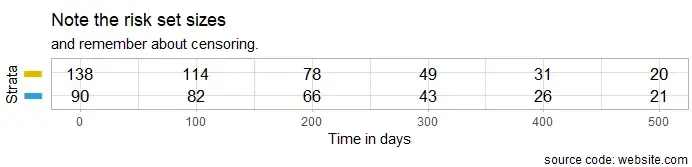
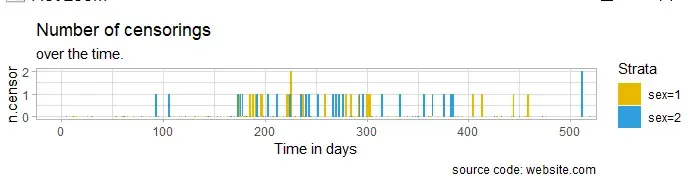
# 绘制删失图标签 ggsurv$ncensor.plot <- ggsurv$ncensor.plot + labs( title = "Number of censorings", subtitle = "over the time.", caption = "source code: website.com" ) ggsurv$ncensor.plot
# 自定义修更改字体大小、类型和颜色 ggsurv <- customize_labels( ggsurv, font.title = c(16, "bold", "darkblue"), font.subtitle = c(15, "bold.italic", "purple"), font.caption = c(14, "plain", "orange"), font.x = c(14, "bold.italic", "red"), font.y = c(14, "bold.italic", "darkred"), font.xtickslab = c(12, "plain", "darkgreen") ) ggsurv
绘制多组生存曲线图
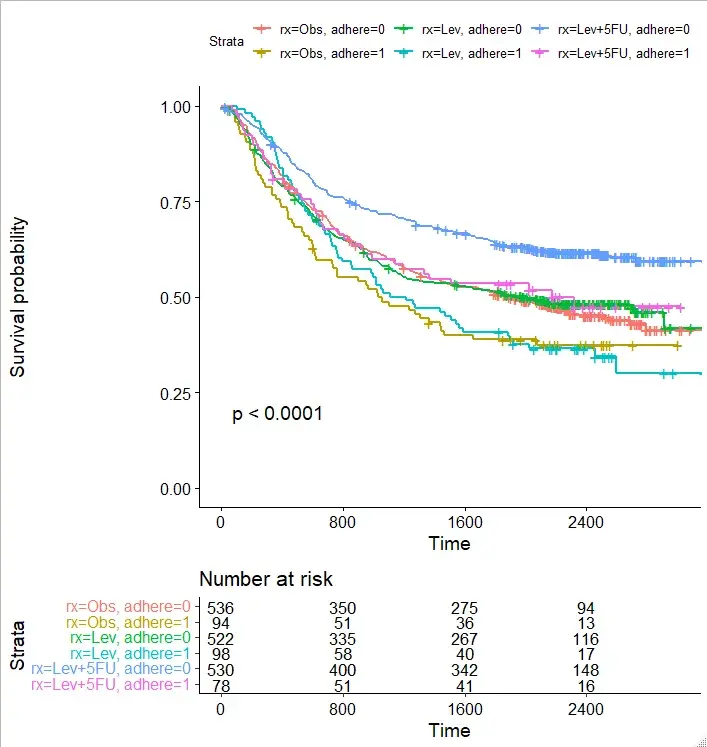
# 多组生存曲线也可以调用相应的函数来绘制 # 再来看看内置数据集colon head(colon) > head(colon) id study rx sex age obstruct perfor adhere 1 1 1 Lev+5FU 1 43 0 0 0 2 1 1 Lev+5FU 1 43 0 0 0 3 2 1 Lev+5FU 1 63 0 0 0 4 2 1 Lev+5FU 1 63 0 0 0 5 3 1 Obs 0 71 0 0 1 6 3 1 Obs 0 71 0 0 1 nodes status differ extent surg node4 time etype 1 5 1 2 3 0 1 1521 2 2 5 1 2 3 0 1 968 1 3 1 0 2 3 0 0 3087 2 4 1 0 2 3 0 0 3087 1 5 7 1 2 2 0 1 963 2 6 7 1 2 2 0 1 542 1 #rx分组 fit2 <- survfit( Surv(time, status) ~ rx + adhere, data = colon ) ggsurvplot(fit2, pval = TRUE, break.time.by = 800, risk.table = TRUE, risk.table.height = 0.5 )
实验外包 想了解更多请关注:https://www.do-gene.com




